Rを用いた偏相関係数の算出
メリークリスマスですね。海外ではHappy Hollidaysと言うんだとか。
さて、投稿していた論文の査読結果が返ってきました。
この研究はサルコペニアの重症化予防に関する研究なのですが、
相関係数も年齢調整したものを出してみたらどうですか?とのこと。。。
回帰分析では年齢や性別で調整した偏回帰係数を算出することが多いのですが、相関係数でも同じようなことはできるのでしょうか?
だいたいRの基本的な統計手法はネットで誰かの手により解説されていますが、偏相関係数に関しては見かけなかったので、調べてみました。
と、いうか、偏相関係数をメインの結果にしている論文を私は見たことがありませんけどね。。。
パッケージはppcorというものです。
偏相関係数に特化していて解説書もコンパクト。
https://cran.r-project.org/web/packages/ppcor/ppcor.pdf
ではでは、フィッシャーのあやめのデータ(iris)を使って解析していきましょう。
Sepal(がく片)Petal(花びら)のそれぞれの長さ(Length)と幅(Width)と
品種(Species):setosa, versicolor, virginica のデータが含まれています。
RはMicrosoft R Open 3.4.2 を使用しています。
パッケージは今回ppcorとpsychが必要です。
knitしたので内容を見ていきましょう!!
Calculate the partial correlation by ppcor.
Urocortin
2017年12月22日
まずはデータを見てみます。
head(iris)## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosa
str(iris)## 'data.frame': 150 obs. of 5 variables:
## $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
describeは非常に便利です。
## 平均、SE、SE、中央値、IQRの計算
describe(iris[,-5],quant=c(.50,.25,.75),ranges=F,skew=F)## vars n mean sd se Q0.5 Q0.25 Q0.75
## Sepal.Length 1 150 5.84 0.83 0.07 5.80 5.1 6.4
## Sepal.Width 2 150 3.06 0.44 0.04 3.00 2.8 3.3
## Petal.Length 3 150 3.76 1.77 0.14 4.35 1.6 5.1
## Petal.Width 4 150 1.20 0.76 0.06 1.30 0.3 1.8
pairsも非常に便利です。相関、散布図、ヒストグラム、密度プロットなどが一目でわかります。(少しかっこ悪いし、相関が有意かどうか分からないので、私は師匠がmodifyしてくれたものを使用しています。)
pairs.panels(iris)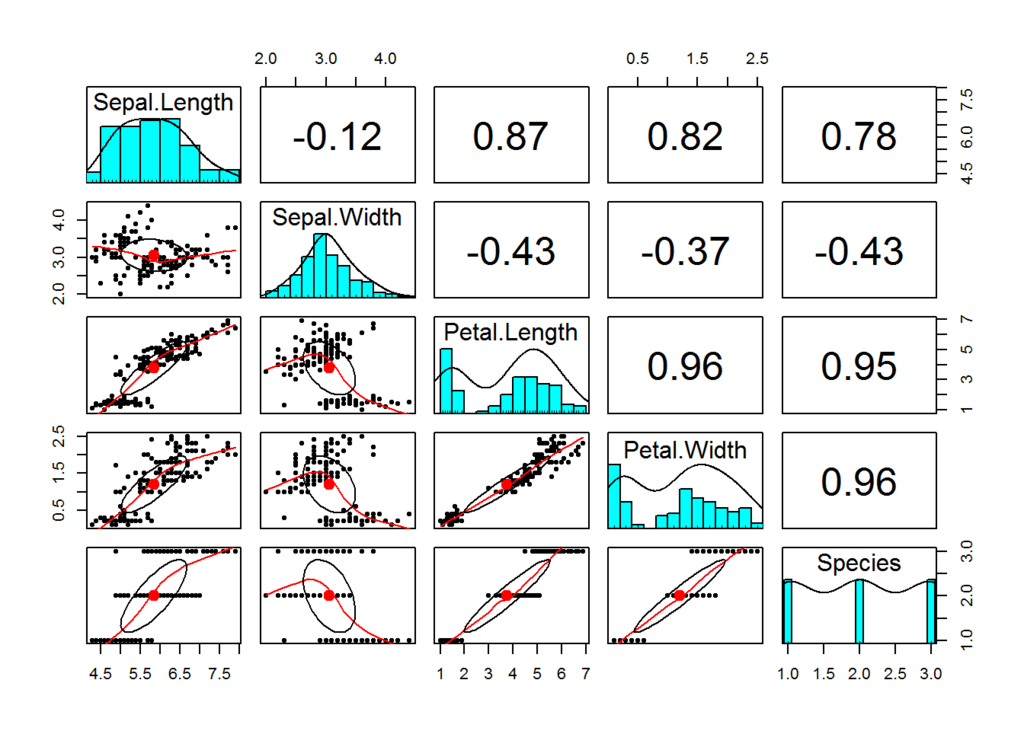
まずは、相関を出してみましょう。
d1 <- iris[,-5]
## 相関行列
### P値は斜めうえの部分が多重検定した分を調整
corr.test(d1)## Call:corr.test(x = d1)
## Correlation matrix
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 1.00 -0.12 0.87 0.82
## Sepal.Width -0.12 1.00 -0.43 -0.37
## Petal.Length 0.87 -0.43 1.00 0.96
## Petal.Width 0.82 -0.37 0.96 1.00
## Sample Size
## [1] 150
## Probability values (Entries above the diagonal are adjusted for multiple tests.)
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 0.00 0.15 0 0
## Sepal.Width 0.15 0.00 0 0
## Petal.Length 0.00 0.00 0 0
## Petal.Width 0.00 0.00 0 0
##
## To see confidence intervals of the correlations, print with the short=FALSE option相関係数とP値が出ます。P値は上の三角が多重検定を調整したP値になります。
単相関でみてみると、細かな情報まで出してくれます。
## 単相関
cor.test(iris$Sepal.Length,iris$Petal.Length, method="pearson") ##
## Pearson's product-moment correlation
##
## data: iris$Sepal.Length and iris$Petal.Length
## t = 21.646, df = 148, p-value < 2.2e-16
## alternative hypothesis: true correlation is not equal to 0
## 95 percent confidence interval:
## 0.8270363 0.9055080
## sample estimates:
## cor
## 0.8717538t値、自由度、P値、帰無仮説が何か、95%信頼区間、相関係数を出力してくれます。
では、実際に偏相関係数を計算してみましょう。めっちゃ簡単です。
## 偏相関係数(他のもので調整した相関係数)とP値
pcor(d1)## $estimate
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 1.0000000 0.6285707 0.7190656 -0.3396174
## Sepal.Width 0.6285707 1.0000000 -0.6152919 0.3526260
## Petal.Length 0.7190656 -0.6152919 1.0000000 0.8707698
## Petal.Width -0.3396174 0.3526260 0.8707698 1.0000000
##
## $p.value
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 0.000000e+00 1.199846e-17 7.656980e-25 2.412876e-05
## Sepal.Width 1.199846e-17 0.000000e+00 8.753029e-17 1.104958e-05
## Petal.Length 7.656980e-25 8.753029e-17 0.000000e+00 7.332477e-47
## Petal.Width 2.412876e-05 1.104958e-05 7.332477e-47 0.000000e+00
##
## $statistic
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 0.000000 9.765380 12.502483 -4.362929
## Sepal.Width 9.765380 0.000000 -9.431189 4.553279
## Petal.Length 12.502483 -9.431189 0.000000 21.398708
## Petal.Width -4.362929 4.553279 21.398708 0.000000
##
## $n
## [1] 150
##
## $gp
## [1] 2
##
## $method
## [1] "pearson"estimateが実際に2者以外の全てで調整した相関係数になります。p.valueがそのP値。
statisticはあまりよくわかりません。(t値?)
nがサンプル数で、gpは与えられた変数がいくつかという事らしいです。
methodは、pearson、kendall、spearmanのいずれも使用できます。
では、2変数の相関を他の1変数で調整してみましょう。
## 第三の変数で調整した二変数間の偏相関係数
pcor.test(iris$Sepal.Length,iris$Petal.Length,iris[,c("Sepal.Width")],method = c("pearson"))## estimate p.value statistic n gp Method
## 1 0.9153894 5.847914e-60 27.56916 150 1 pearson出力は同じようにでてきます。
私のように、AとBの年齢調整した相関を出したい!という時はこのようにすればOKです。
ちなみに、調整変数を増やすこともできます。
## 調整するものを増やすことも可能(Factorはダメ)
pcor.test(iris$Sepal.Length,iris$Petal.Length,iris[,c("Sepal.Width","Petal.Width")],method = c("pearson"))## estimate p.value statistic n gp Method
## 1 0.7190656 7.65698e-25 12.50248 150 2 pearson変数にfactorは用いれません!あと、欠損値もダメです。
意外と使える範囲は狭そう。。。
今回は偏相関係数の使用方法を書きましたが、前述したとおり、相関係数を論文のメインのアウトカムに持ってくることはあまりしませんよね。偏相関も論文ではほとんど見たことがありません。
再解析してみると、年齢調整してもあまり大きくは結果が変わりませんでしたし、limitationのところに、Additional analyses adjusting for age did not alter the results.とでも追加しておきましょうかね。
今日はこのへんで。間違ってたら教えてください。